【Data Lab】哈爾濱N居疫情傳播動態數據分析
新冠肺炎在全球越演越烈,但這兩天哈爾濱的一個社區傳播卻引發了華文圈的高度矚目。
這真的很難簡單說,總之就是一名男子韓某從美國回到哈爾濱,在居家檢疫期間劈了鄰居曹女,而曹女又與男友李男發生性行為,而曹女與母親王女有居住事實,而王女又劈了老情人郭某,王女又與郭某和陳姓朋友吃飯,然後這位陳姓朋友發病後疫情就一發不可收拾。然後疫調單位從一發不可收拾的疫情中抽絲剝繭,發現陳姓朋友往上王女與郭某兩人可以向上溯源,但王女與郭某等人又拒不吐實,經過曉以大義後,才追出韓某、曹女、李男複雜的關係。
相信看到這裡,大家頭都昏了。比起其他疫情傳播案例相對不透明,由於這個事件非常驚爆,媒體與公眾號有呈現完整的傳播路徑,但各媒體的傳播路徑圖都做得有點土炮:
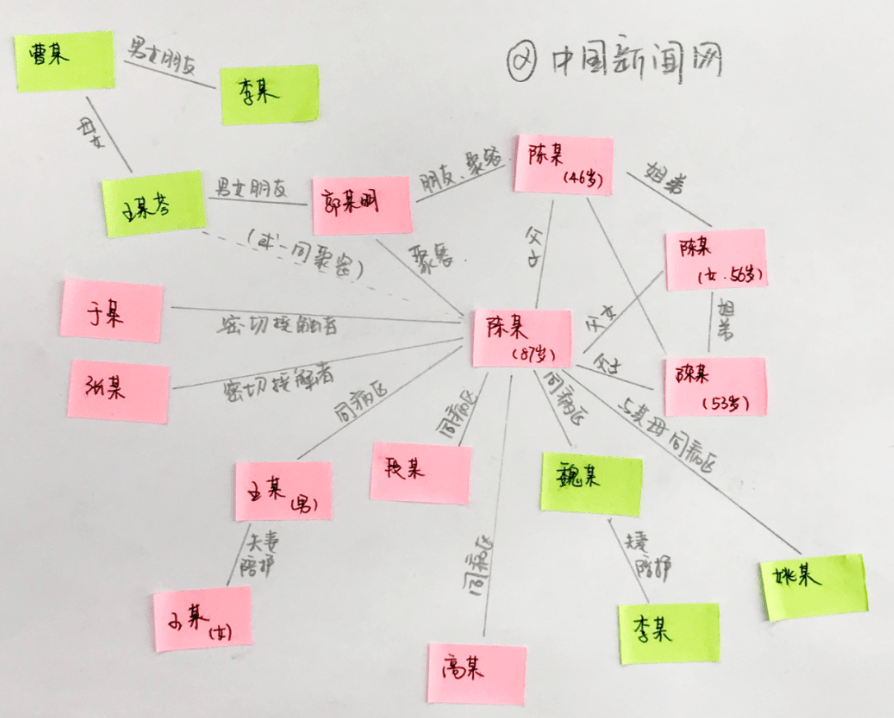
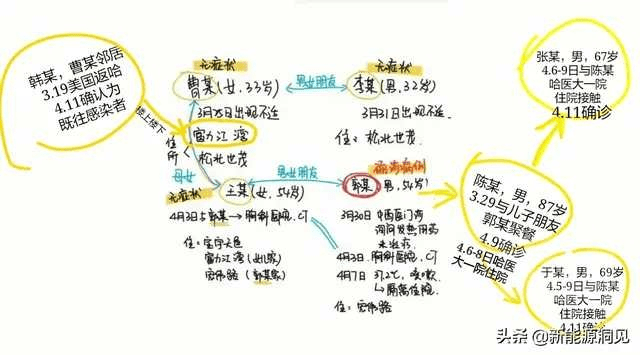
這樣實在無法讓我們清楚看到整個疫情散播由小而大的過程,而且也無法進行更深入的數據分析,比方啪啪啪的疫情傳播威力比較各種傳播模式的程度為何,這牽涉到嚴肅的防疫漏洞問題。
所以我這裡用R的sna、tsna、ndtv等套件,來做一次完整的疫情傳播數據該怎麼紀錄、呈現及分析。下面就是動態圖的呈現結果,不是清楚多了嗎?:(t為天數,可手動拉時間軸,也可按play自動跑時間看發展狀況。)
在這個全球疫情仍然嚴峻的時候,這個案例能初步展示一個疫情傳播的數據處理模式,紀錄表格格式做好,紀錄一次,可視化、社會網絡分析都能一次到位,對於疫調單位未來進行疫情調查及公眾溝通,有莫大的幫助(誤)。
1. 疫情紀錄
從疫情傳播該如何紀錄來看,傳染病的模式一傳一或一傳多,如A → B,B → C,B → D等等,其實就是social network的格式,所以可以建立一個有source、target及其他相關資料的表格,從最簡單的疫調需求來看,可以用一張表格進行完整的紀錄,表格欄位應該是這樣:
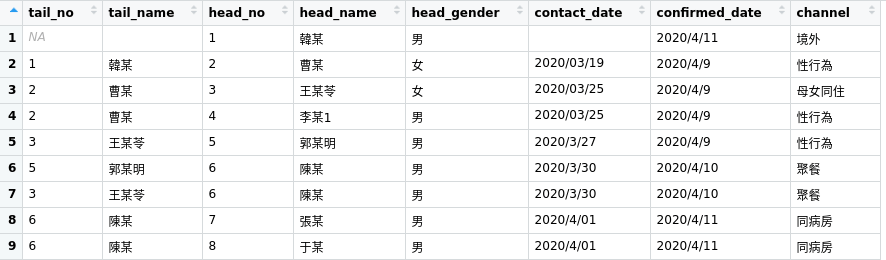
可以下載我以目前揭露的疫情所紀錄的測試檔案,並用R讀入:
harbin <- read.csv("data/harbin_sn.csv", stringsAsFactors = FALSE)
欄位說明如下:
- tail_no:傳播者編號
- tail_name:傳播者姓名
- head_no:被感染者編號
- head_name:被感染者姓名(李某1的其它女友不知人數,這裡用兩名示意。之後的醫護人員感染是媒體報導,並無準確感染人數,這裡以增加11名醫護人員示意)
- head_gender:被感染者性別
- contact_date:傳播者、被傳播者接觸日期(不是準確日期,我是按照新聞進行推算,我又不是疫調單位)
- confirmed_date:確診日期
- channel:感染途徑
當然疫調單位可以自行輸入更多所需要的欄位,例如contact_date可以是一段期間,國籍、傳播地點等等。
2. 疫情數據可視化呈現
要以tsna、ndtv套件做出疫情散播時間動態圖,需要將上面的檔案轉成四個表格:
- nodes:各個節點基本資料
- edges:連結模式資料
- nodes_dy:節點出現、結束時間
- edges_dy:連結出現、結束時間
程式碼如下:
library(sna)
library(tsna)
library(ndtv)
library(dplyr)
library(tidyr)
#edges
edges <- harbin %>%
select(one_of("tail_no", "head_no"))
edges <- edges[-1,]
colnames(edges) <- c("tail", "head")
#nodes
nodes <- harbin %>%
select(1:5, 8)
#nodes重複測試
nodes_tail <- nodes %>%
select(one_of("tail_no")) %>%
distinct()
nodes_head <- nodes %>%
select(one_of("head_no")) %>%
distinct()
setdiff(nodes_tail$tail_no, nodes_head$head_no)
#nodes單一化
nodes <- nodes[duplicated(nodes$head_no) == FALSE, 3:6]
colnames(nodes) <- c("vertex.id", "name", "gender", "channel")
#nodes_dy
nodes_dy <- harbin[duplicated(harbin$head_no) == FALSE, c(3, 6)]
nodes_dy$end <- c("2020/4/10") #以4月10日作為動態圖結束日
nodes_dy$contact_date[1] <- "2020/3/19"
nodes_dy$contact_date <- as.POSIXct(nodes_dy$contact_date)
nodes_dy$end <- as.POSIXct(nodes_dy$end)
#3月19日為第一天,4月10日為最後一天,進行計算每一天是第幾天
nodes_dy$onset <- ((nodes_dy$contact_date - as.POSIXct("2020-03-19")) / (24 * 60 * 60)) + 1
nodes_dy$terminus <- (as.POSIXct("2020-04-10") - as.POSIXct("2020-03-19"))
nodes_dy <- nodes_dy[, c(4, 5, 1)]
colnames(nodes_dy)[3] <- c("vertex.id")
#ndtv需要讀取回存的csv檔案
write.csv(nodes_dy, "nodes_dy.csv", row.names = FALSE)
nodes_dy <- read.csv("nodes_dy.csv", stringsAsFactors = FALSE)
#edges_dy
edges_dy <- harbin[, c(1, 3, 6)]
edges_dy$end <- c("2020/4/10")
edges_dy <- edges_dy[-1,]
edges_dy$contact_date <- as.POSIXct(edges_dy$contact_date)
edges_dy$end <- as.POSIXct(edges_dy$end)
#3月19日為第一天,4月10日為最後一天,進行計算每一天是第幾天
edges_dy$onset <- ((edges_dy$contact_date - as.POSIXct("2020-03-19")) / (24 * 60 * 60)) + 1
edges_dy$terminus <- (as.POSIXct("2020-04-10") - as.POSIXct("2020-03-19"))
edges_dy <- edges_dy[, c(5, 6, 1, 2)]
colnames(edges_dy)[3:4] <- c("tail", "head")
#ndtv需要讀取回存的csv檔案
write.csv(edges_dy, "edges_dy.csv", row.names = FALSE)
edges_dy <- read.csv("edges_dy.csv", stringsAsFactors = FALSE)
四個檔案好了之後,就能開始製作動態圖:
#基礎社會網路
harbin_network <- network(
edges,
vertex.attr = nodes,
vertex.attrnames = c("vertex.id", "name", "gender", "channel"),
directed = TRUE,
bipartite = FALSE
)
plot_position <- plot(harbin_network) #除存節點位置,否則動態圖節點會亂跳
繪製ndtv動態圖:
harbin_dynamic <- networkDynamic(
harbin_network,
edge.spells = edges_dy,
vertex.spells = nodes_dy
)
network::set.vertex.attribute(harbin_dynamic, 'x', plot_position[,1])
network::set.vertex.attribute(harbin_dynamic, 'y', plot_position[,2])
reconcile.vertex.activity(harbin_dynamic, mode = "match.to.edges")
compute.animation(
harbin_dynamic,
animation.mode = "useAttribute",
slice.par = list(
start = 0,
end = 23,
interval = 2,
aggregate.dur = 1,
rule = "latest"
)
)
render.d3movie(
harbin_dynamic,
usearrows = TRUE,
displaylabels = TRUE,
label = paste(harbin_dynamic%v%"vertex.id",
harbin_dynamic%v%"name", "途徑:", harbin_dynamic%v%"channel")
)
瀏覽器上就會出現下面的動態圖了:
3. 疫情分析
從圖上可以簡單看得出來,王女又與郭某之前劈來劈去的事件,其實都蠻單一的,重點還是後來左邊的那一陀,從社會網路分析來看,我們從中心度等指數來看一下:
harbin_anal <- data.frame(nodes, infocent = infocent(harbin_network), degree = sna::degree(harbin_network), betweenness = flowbet(harbin_network)) harbin_anal <- arrange(harbin_anal, desc(degree))
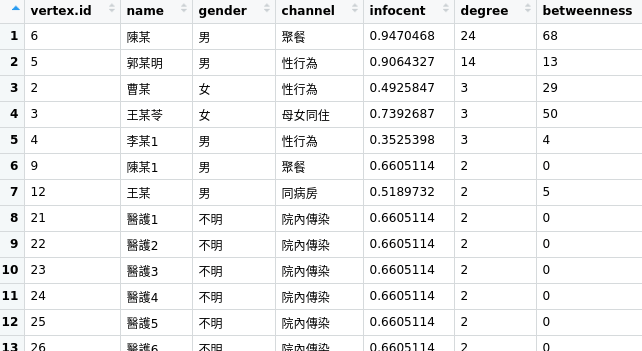
可以明顯看出,從information centrality、degree、betweenness等幾個指數來看,6號的陳某都是傳播的中心。
再來看information centrality的變化:
dynamicBetweenness <- tSnaStats( harbin_dynamic, snafun = "centralization", start = 0, end = 23, time.interval = 2, aggregate.dur = 1, FUN = "infocent" ) plot(dynamicBetweenness)
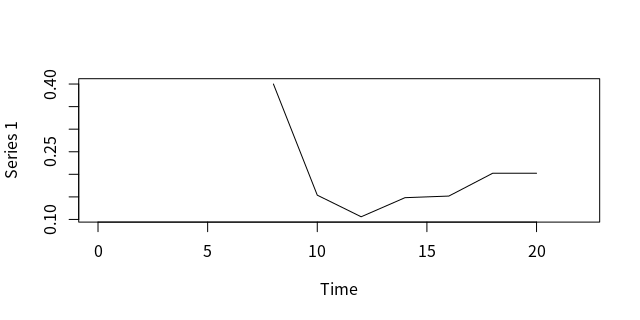
可以看得出來一開始網絡只有少數幾個點的時候,中心度很高,在3月底之後節點變多,中心度下降,卻因為6號陳某在醫院傳播疫情,拉高中心度。
4. 結論
所以我們從上面的可視化和分析做出結論:
- 我在這裡嚴肅呼籲,從數據分析來看,哈爾濱N居疫情傳播真正該注意的是院內感染!初始階段的劈來劈去雖然很狗血,但不要被轉移焦點!
- 社會網絡分析的動態化,不僅能應用在疫情,商業交易、社群網路等人與人之間的關係行為,以時間動態呈現,會比靜態處理分析更多的東西。
- 社會網絡格式進行疫情的紀錄,可以有可視化、網絡數據分析等多重優點,如果在端點、連線之間作一些分類處理,能夠分析的數據更多,接觸日期與確診日期也能分別作處理看疫情可能哪裡出漏洞。雖然目前台灣的傳播節點及路徑都非常短,但應該還能看出一些有意思的東西。
喜欢我的作品吗?别忘了给予支持与赞赏,让我知道在创作的路上有你陪伴,一起延续这份热忱!
